 © Isabelle Domaizon
© Isabelle Domaizon
Quanti-Inventory
Consolidation des méthodes quantitatives pour le suivi des assemblages piscicoles par l’ADNe: validation du choix méthodologique
SECTIONSurveillance innovante |
PERIODEJanvier 2024 - Janvier 2025 |
|
FINANCEMENTPôle ECLA |
ZONE(S) D'ÉTUDEFrance hexagonale |
|
PARTENAIRESUniversité Savoie Mont Blanc, INRAE, UMR CARRTEL, OLA 74200 Thonon-les-Bains, FranceService EcoAqua, DRAS, OFB, Aix-en-Provence, France Aix Marseille Univ., INRAE, RECOVER, 13 182 Aix-en-Provence, France Jean Guillard INRAE, UR-Riverly, Centre de Lyon-Villeurbanne, Villeurbanne Cedex, France |
COORDINATEUR(S)I. Domaizon |
Description du Projet
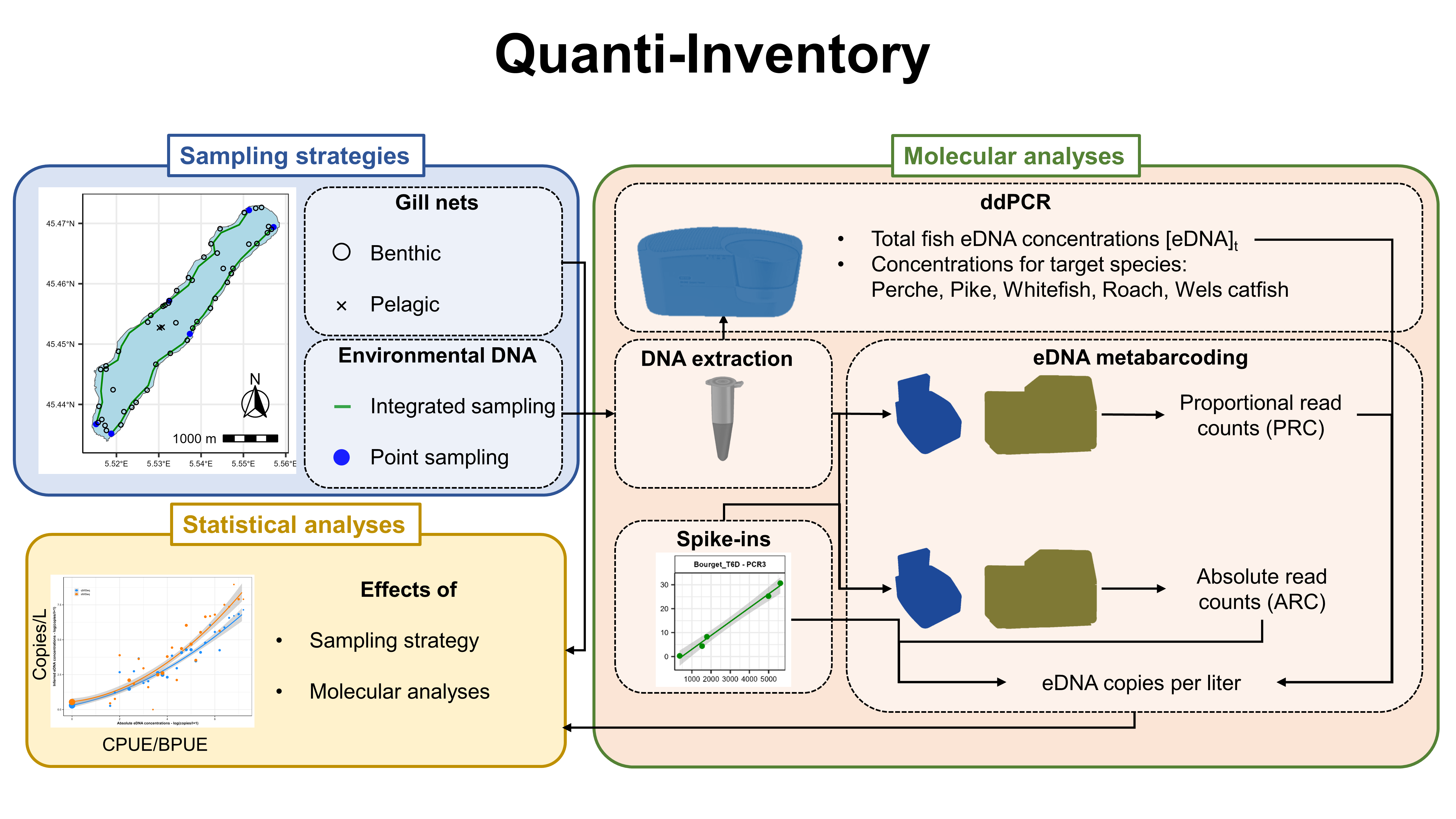
Contexte du Projet :Ce projet s’inscrit dans la continuité des travaux visant à extraire une information quantitative à partir des suivis par ADN environnemental (ADNe) pour les communautés piscicoles. Les attentes sont fortes pour aboutir à des données ADNe reflétant l’abondance-biomasse des espèces. A cette fin deux stratégies sont proposées : l’une combine le métabarcoding et la PCR quantitative (Pont et al 2022), l’autre repose sur l’ajout de standards internes (Ushio et al 2018). Toutefois, une comparaison rigoureuse de ces deux approches doit être mener afin d’évaluer leur robustesse et leur transférabilité.
Objectifs du Projet :L’objectif est de comparer deux approches transformant les séquences ADN issues du metabarcoding en nombre de copies par espèce, afin d’améliorer l’approximation de l’abondance et de la biomasse des assemblages piscicoles. Ce travail vise (i) à élaborer un protocole opérationnel, combinable avec des techniques non invasives comme l’hydroacoustique, pour un suivi efficace des communautés piscicoles lacustres, et (ii) plus largement, à renforcer les informations quantitatives du metabarcoding ADNe pour leur intégration dans les programmes nationaux de surveillance écologique.
Résultat du Projet
Pour aller plus loin
Liens Utiles :
https://doi.org/10.1101/2025.09.05.674411
Contact : isabelle.domaizon[at]inrae.fr